Świńska grypa jest bardzo zaraźliwą infekcją wirusową układu oddechowego świń, która stała się enzootyczna na obszarach o dużej gęstości lokalizacji gospodarstw trzody chlewnej. Może to mieć istotny wpływ na gospodarkę w dotkniętcyh stadach i może stanowić zagrożenie dla populacji ludzkiej, poniweaż wirus grypy świń (SIV) jest patogenem zoonotycznym. Zdobywana na bieżąco wiedza na temat odmian SIV jest niezbędna dla zapobiegania i zwalczania tej choroby u świń i może stanowić podstawę do oceny ryzyka transmisji międzygatunkowej, w kontekście przygotowania się na wypadek pandemii.
SIV należy do rodzaju Influenzavirus A , rodzina Orthomyxoviridae. Ich genom zawiera osiem unikatowych segmentów jednoniciowego RNA o ujemnej polaryzacji. Są podtypowane zgodnie z ich głównymi antygenowymi determinantami powierzchni glikoprotein hemaglutyniny (HA) oraz neuraminidazy (NA). Pojawienie się nowego SIV u świni może wystąpić za pośrednictwem transmisji wirusa z innych gatunków, zmiany antygenowej głównych antygenów wirusowych przez mutacje (dryf) lub rearanżacje genetyczne (wymiana genów między dwoma lub więcej wirusami). Skok antygenowy (shift) obserwuje się, gdy geny HA i NA są zamienione podczas rearanżacji.
Obecnie wśród świń na całym świecie krążą trzy podtypy wirusa (H1N1, H3N2, H1N2) jednak ich pochodzenie jest różne w zależności od regionu i kilku linii genetycznych mogących istnieć w obrębie podtypu. W Europie dominujące H1N1 są wyłącznie pochodzenia ptasiego, wprowadzone od ptactwa wodnego do trzody chlewnej w 1979 roku i zwana dalej "podobna do ptasiej grypa świń H1N1" (H1avN1). Wirus grypy świń H3N2 został wprowadzony do Europy krótko po pandemii w Hongkongu w 1968 roku, ale rosprzestrzenił się po reasortacji z wirusem H1avN1, nabywając 6 wewnętrznych genów od tego ostatniego. Ten pochodzący od człowieka wirus grypy świń H3N2 jest obecnie dominującym genotypem wirusa H3N2 u świń europejskich. W 1994 r. H3N2 uzyskał gen HA wirusa ludzkiej grypy H1N1 z 1980 roku, w wyniku czego obecnie dominującym jest "podobny do ludzkiego reasortant wirusa grypy świń H1N2" (H1huN2). Te trzy SIV wspólnie krążą przez wiele lat, ale nowe reasortanty wirusów między tymi enzootycznymi (lub między SIV i sezonowymi wirusami ludzkimi) wykrywa się od 2000 roku. Przesunięcie antygenowe wystąpiło poprzez pojawienie się reasortantów drugiej generacji, takich jak rH1avN2 i rH1huN1, ale takie izolacje zazwyczaj były sporadyczne i tylko jeden taki wirus rH1avN2 zastąpił powszechnie wystepujący H1huN2 w danym regionie. Ewolucję wirusów zachodzącą poprzez antygenowy dryf udało się ograniczyć u świń europejskich w ostatnich latach, bez względu na podtyp. Tak więc sytuacja była dość stabilna w europejskich stad trzody chlewnej, aż do pojawienia się w 2009 roku z pandemii wirusa H1N1 (H1N1pdm) pochodzenia świńskiego.

Wirus influenzy świń wykryty u świń europejskich w 2014 roku
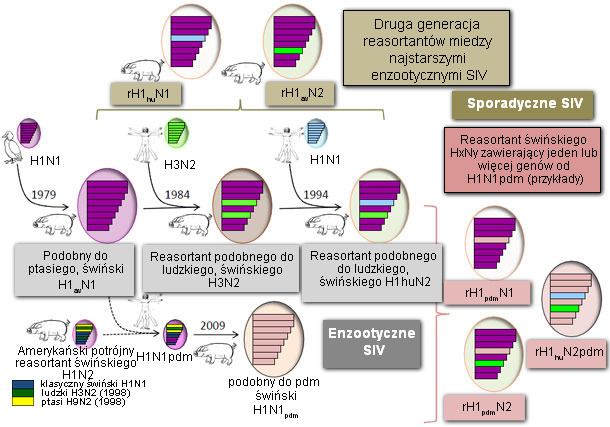
H1N1pdm został wygenerowany z rearanżacji pomiędzy amerykańskim potrójnym reasortantem SIV (wykazującego "typowe" geny HA pochdzące od świń, drobiu i ludzi) i wirusem H1avN1 z których najpierw nabył geny NA i macierzy (M). Świnie okazały sę być bardzo podatne na tego ludzkiego wirusa pochodzenia świńskiego. Bardzo szybko po jego pojawieniu się u człowieka odnotowano liczne ogniska zakażeń zoonotycznych całym świecie. W większości głównych krajów produkujących, w tym w Europie wirus dostosował się do świń i kontynuował rozprzestrzenianie się wirusa u świń. Wkrótce po transmisji międzygatunkowej, wykryto także kilka reasortantów między starym, enzootycznym SIV i H1N1pdm.
Cztery linie wirusów, wyraźnie odróżniające się od HA, obecnie wspólnie krążących wśród świń europejskich, mogą być uważane za wirusy enzootyczne, nawet jeśli ich względna częstość i poziom zachorowalności wahają się omiędzy krajami. Dominującą linią jest wciąż najstarszy H1avN1, następnie H1huN2, H1N1pdm i H3N2. Podtypu H3N2 brakuje w niektórych obszarach geograficznych, podczas gdy nadal jest powszechny w innych. Co ciekawe, obszary wolne od H3N2 to te, które wykazują najwyższą częstotliwość występowania wirusów H1N2 (H1huN2 lub rH1avN2) . Reasortanty H1N2 zawierające geny od wirusa H1N1pdm wykrywa się coraz częściej na przestrzeni ostatnich dwóch lat, co oznacza, że niektóre z nich mogą również być na stałe w stadach trzody chlewnej. Ilustrują one rosnącą różnorodność i złożoność genetyczną SIV europejskich i potencjalnie zapewniają materiał do infekcji zoonotycznych.
Zdecydowanie potrzebne jest kontynuowanie nadzoru i aktualizowanie wiedzy na temat wirusów, dzielenie się tą wiedzą między rolnikami, weterynarzami, naukowcami i innymi zainteresowanymi stronami.


