Nie tak nowe wyzwania związane z wirusem PRRS
Nie jest nowością, że wirus zespołu rozrodczo-oddechowego świń (PRRSV) ma istotne cechy, które przyczyniają się do ciągłego nawrotu choroby w stadach świń, co prowadzi do znacznych problemów zdrowotnych zwierząt i strat ekonomicznych. Jedną z tych cech jest jego szybka zdolność do mutacji i potencjalnego rozwoju choroby i/lub zarażania zwierząt, które wcześniej były "odporne" na inny wariant PRRSV. Z tego powodu, aby zrozumieć "typ" PRRSV obecny w gospodarstwie - niezależnie od tego, czy jest to wirus z nowego ogniska, czy wirus "rezydentny" - bardzo powszechne stało się analizowanie genomu PRRSV.
Zadanie to jest zwykle wykonywane poprzez sekwencjonowanie ważnej części PRRSV zwanej genem ORF5; aby zrozumieć, gdzie wirus pasuje ewolucyjnie i jak wypada w porównaniu z poprzednimi wirusami wykrytymi w danym miejscu lub w okolicy/regionie.

Informacje o sekwencjonowaniu PRRSV były szeroko wykorzystywane w terenie, w celu
- zrozumienia, który wirus powoduje problemy
- wskazówek dotyczących źródła ognisk choroby
- rozróżnienia między szczepami szczepionkowymi a szczepami "rezydującymi"/nowo wprowadzonymi
- wyboru szczepionki itp.
Ostatnio do klasyfikacji wirusów coraz częściej stosuje się klasyfikację " linii genetycznych". Metoda ta została opracowana około 2010 roku (Shi et al., 2010), kiedy PRRSV został podzielony na 9 odrębnych linii genetycznych; a następnie udoskonalona, gdy PRRSV został dalej podzielony na podlinie.
Słoń w pokoju: czy sekwencjonowanie jednej próbki jest wystarczająco dobre?
Sekwencjonowanie PRRS zwykle nie jest tanie, dlatego jest częściej wykonywane w "szczególnych sytuacjach", a nie rutynowo (np. podczas nowych ognisk, po inokulacji wirusem itp.). Ponadto w takich przypadkach tylko jedna próbka lub pula próbek (z wielu powszechnie pobieranych w gospodarstwie) jest wykorzystywana do sekwencjonowania w zdecydowanej większości przypadków diagnostycznych.
Wynika z tego, że w wielu przypadkach lekarze weterynarii, producenci i inny personel zajmujący się zdrowiem zwierząt powszechnie wykorzystują informacje o pojedynczej sekwencji do wyciągania ważnych wniosków dotyczących źródła wirusa (zwłaszcza w przypadku nowych ognisk), kierowania dochodzeniami w sprawie ognisk i informowania o przyszłych interwencjach.
W naszym badaniu chcieliśmy potwierdzić, że gdybyśmy sekwencjonowali wiele próbek pobranych w ciągu tego samego dnia, wszystkie próbki dałyby tę samą sekwencję lub sekwencje, które byłyby "wystarczająco zbliżone", aby można je było uznać za co najmniej tę samą linię wirusa. Chcieliśmy również spróbować sekwencjonowania całego genomu (WGS) wraz z sekwencjonowaniem ORF5, aby sprawdzić, czy uzyskamy więcej informacji. WGS może okazać się korzystne, ponieważ dostarcza informacji na temat całego wirusa, podczas gdy ORF5 był wykorzystywany w przeszłości, ale mówi nam tylko o wycinku jego historii. Ponieważ jednak jest to wciąż stosunkowo nowe rozwiązanie i może być kosztowne (w Stanach Zjednoczonych kosztuje około trzy razy więcej niż sekwencjonowanie ORF5), wciąż uczymy się, jak je interpretować, a nasza "baza danych" do porównania jest znacznie mniejsza w porównaniu z ORF5.
Podejrzewaliśmy, że biorąc pod uwagę szybkie tempo mutacji PRRS i duże rozmiary nowoczesnych zakładów hodowli trzody chlewnej, możemy znaleźć wiele linii PRRS przy jednorazowym przypadku pobierania próbek, co byłoby niepokojące. Graficzne podsumowanie naszego uzasadnienia badania znajduje się na ryc. 1.
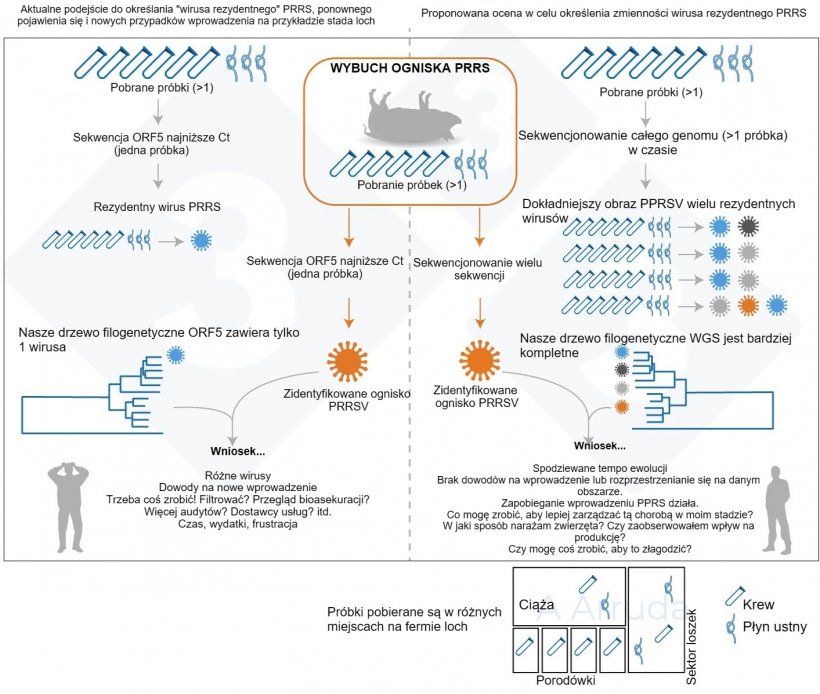
Badanie, ile szczepów PRRS możemy znaleźć na naszych farmach
Do badania włączyliśmy pięć gospodarstw, 3 stada hodowlane i 2 stada tuczowe; próbki pobierano co miesiąc przez około rok, wykorzystując różne rodzaje próbek (zeskrobiny z migdałków, płyny ustne, płyny po kastracji i obcinaniu ogonów). Zawsze pobieraliśmy próbki z tych samych miejsc w stadzie, które były rozmieszczone przestrzennie w całym obiekcie, aby uzyskać jak największą reprezentację całego terenu. Co miesiąc pobieraliśmy do 16 próbek z każdego miejsca, a wszystkie z nich zostały przetestowane przy użyciu ilościowej reakcji PCR; a następnie dodatnie próbki zostały poddane sekwencjonowaniu ORF5.
Czego się dowiedzieliśmy?
Naszym najważniejszym odkryciem było to, że w warunkach terenowych byliśmy w stanie wykryć do trzech różnych linii PRRSV podczas pojedynczego pobierania próbek w stadach hodowlanych i do dwóch w stadach tuczowych. Zostało to pokazane na Ryc. 2.
Ryc 2.Podlinie PRRSV wykryte w badanych fermach (1-5) przez cały okres pobierania próbek (w miesiącach) dla różnych typów próbek.
| Kolejne pobierania próbek | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Ferma | Typ próbki | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 1 | Płyn pokastracyjny i po obc. ogonów | L1H | L1H | ||||||||||
| Zeskrobina z migdałków | |||||||||||||
| 2 | Płyn pokastracyjny i po obc. ogonów | L1H | L1H | L1A | L1H | L1H | |||||||
| L1H | |||||||||||||
| L8† | |||||||||||||
| Zeskrobina z migdałków | L1H | ||||||||||||
| 3 | Płyn pokastracyjny i po obc. ogonów | L1H | L1H | L1H | L1H | ||||||||
| Zeskrobina z migdałków | |||||||||||||
| 4 | Płyn ustny | L5† | L1A | ||||||||||
| L5† | |||||||||||||
| Zeskrobina z migdałków | L1A | L5† | L5† | L1A | |||||||||
| L5† | |||||||||||||
| 5 | Płyn ustny | L1A | L1A | L5† | |||||||||
| Zeskrobina z migdałków | L1A | L1A | |||||||||||
Odnotowaliśmy również, że w przypadku naszego badania próbki płynu ustnego były bardzo trudne do sekwencjonowania, podczas gdy płyny pokastracyjne i po obcinaniu ogonów, najłatwiejsze. Biorąc pod uwagę, że obie próbki są "złożone", można to potencjalnie wyjaśnić ogólnie wyższymi wartościami Ct, wskazującymi na mniejszą ilość wirusa w płynach ustnych w porównaniu z ww. płynami. Próbowaliśmy podjąć próbę wykonania WGS, ale ostatecznie nie byliśmy w stanie uzyskać pełnych sekwencji całego genomu z żadnej z naszych próbek. Podejrzewamy, że może to być spowodowane rodzajem próbek, które wykorzystaliśmy, ale potrzebne są dalsze badania na ten temat.
Wyniki te pokazują, jak ważne jest rozważenie sekwencjonowania wielu próbek przy podejmowaniu ważnych decyzji, nawet jeśli zdajemy sobie sprawę, że wiąże się to z wyższymi kosztami.
Podziękowania
National Pork Board zapewnił finansowanie tego projektu. Chcielibyśmy podziękować lekarzom weterynarii i producentom, którzy odegrali kluczową rolę w zbieraniu próbek.





